生命科学部 三嶋 雄一郎 准教授と理化学研究所などの研究グループが、細胞の中でリボソームが交通渋滞を起こす要因を明らかにしました
2020.05.07
研究概要
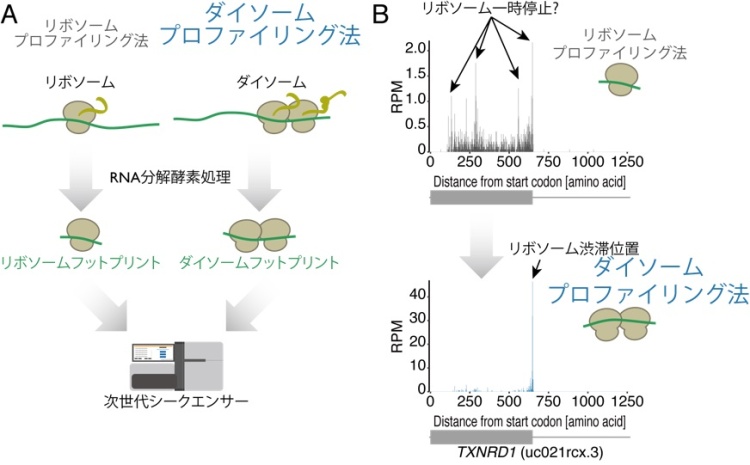
「セントラルドグマ[1]」における最後のステップである翻訳は、メッセンジャーRNA(mRNA)[2]の遺伝情報を最終産物のタンパク質に転換するプロセスであり、さまざまな因子によって制御されています。翻訳の主役を担うリボソーム[3]は一定の速度でmRNAを翻訳してタンパク質を作るのでなく、そのスピードは多様な因子に影響されます。そのため、リボソームは一時停止し、mRNAの上で「リボソーム渋滞」を引き起こすことがあります。一時停止しても翻訳を再開するならば、細胞のホメオスタシスに影響を与えませんが、翻訳が完全に止まると、リボソームは不完全なペプチドを生産して細胞に毒性をもたらす恐れがあります。これを防ぐために、細胞は複数の経路でリボソームの翻訳速度を監視しています。これまで、適切な技術が存在しなかったために、細胞中のどのmRNAのどの位置でリボソーム渋滞が起きるのか、またその原因などについてはよく分かっていませんでした。
今回、共同研究グループは、リボソームの渋滞時に生じるダイソーム(衝突した2個のリボソーム)から得られるmRNA断片を次世代シークエンサー[4]を用いて読み取るダイソームプロファイリング法(図A)を開発し、渋滞の位置をヒト細胞と熱帯魚ゼブラフィッシュの全ゲノムで特定しました(図B)。さらに、渋滞は複数の特定アミノ酸配列や終止コドン[5]などで起きること、渋滞したリボソームから合成される新生ペプチドの一部は品質管理機構により分解されることなどを明らかにしました。
本研究成果は、翻訳そのものに関する基礎研究はもとより、リボソーム渋滞が原因で発症する疾患(神経変性疾患など)の理解に貢献すると期待できます。
詳細は理化学研究所のプレスリリースをご覧ください。
用語解説
[1]セントラルドグマ
細胞内の遺伝情報が、ゲノムDNAの中の遺伝子配列からmRNAに「転写」され、さらに、そのコーディング配列からタンパク質へと「翻訳」されるという、生命に普遍的な決まり(ドグマ)のこと。
[2]メッセンジャーRNA(mRNA)
メッセンジャーRNAは、タンパク質のアミノ酸の並び方の情報(コドン)を持つRNAである。リボソームによってそのコドンが読み取られ、タンパク質が合成される。
[3]リボソーム
リボソームは、メッセンジャーRNA(mRNA)の翻訳を行なう(遺伝情報からタンパク質を合成する)場所。真核生物では、40Sの小サブユニットと60Sの大サブユニットからなる。リボソームの渋滞は、翻訳を停止したリボソームに次のリボソームが衝突して起こる現象。
[4]次世代シーケンサー
DNAの塩基配列を決定するための装置で、DNA断片の塩基配列を同時並行的に、より高速高精度に決定することができる。
[5]コドン、終止コドン
タンパク質の中のアミノ酸の並び方は、そのタンパク質の遺伝子(DNA)の中の塩基配列に対応している。3つの塩基がひとまとまりになって一つのアミノ酸に対応しており、この3塩基の並びを「コドン」と呼ぶ。64通りのコドンが存在するが、そのうちの61種類のコドンがアミノ酸を意味しており、例えばTTTはフェニルアラニンである。残り三つのコドン(TAA、TGA、TAG)はタンパク質合成終了を意味する「終止コドン」であり、遺伝子の最後に現れる。掲載論文
| 論文タイトル | Genome-wide survey of ribosome collision |
|---|---|
| 掲載誌 | Cell Reports |
| DOI | 10.1016/j.celrep.2020.107610 |
| 研究体制 | 理化学研究所、京都産業大学、東北大学、奈良先端科学技術大学院大学の共同研究 |
| 著者 | Peixun Han*1、Yuichi Shichino*1、Tilman Schneider-Poetsch*1、Mari Mito*1、Satoshi Hashimoto*3、 Tsuyoshi Udagawa*3、Kenji Kohno*4、Minoru Yoshida*1、Yuichiro Mishima*2、Toshifumi Inada*3、 Shintaro Iwasaki*1 |
*1理化学研究所、*2京都産業大学、*3東北大学、*4奈良先端科学技術大学院大学
