水素イオンの流れを使ってATP合成酵素が回転する仕組みを解明
2024.11.20
京都産業大学生命科学部の横山謙教授らの研究グループは、クライオ電子顕微鏡法と分子動力学シミュレーションを用いて、好熱菌由来のATP合成酵素の詳細な構造とその回転メカニズムを明らかにしました。本研究成果は、代謝や疾病にも関係する重要な創薬ターゲットタンパク質であるATP合成酵素の分子機構と詳細な構造解明であり、将来的な医薬応用につながることが期待できます。
リリース日:2024-11-20
発表論文
(原核生物Voモーターのプロトン駆動力による回転メカニズム)
概要
ATP(アデノシン三リン酸)は、生命にとって最も重要なエネルギー物質であり、細胞のミトコンドリアに存在するATP合成酵素という巨大なタンパク質によって合成されます。この酵素は、生体膜を横断する水素イオンの流れを回転力に変換し、その力でATPを生成します。これまで、水素イオンの流れがどのようにしてATP合成酵素を回転させるのか、その詳細な分子メカニズムは不明でした。しかし、本研究では、ATP合成酵素の回転力を発生させる膜内在部分(Vo部分)の詳細な構造をクライオ電子顕微鏡で解明し、回転力発生に重要な役割を果たすアミノ酸残基の向きを特定しました。さらに、Vo部分の構造を基に分子動力学シミュレーションを行い、水素イオンの流れによる回転メカニズムを解析しました。その結果、回転リングにある特定のアミノ酸残基の荷電状態の変化が回転力発生に重要であることがわかりました。ATP合成酵素は、ヒトから細菌に至る幅広い生物界に存在するタンパク質です。本研究で特定されたアミノ酸残基はすべてのATP合成酵素にほぼ保存されており、この発見は生命がATPを合成する最も重要な仕組みの解明に貢献するものです。本成果は、2024年11月20日(水)19:00(日本時間)に国際学術誌「Nature Communications」にてオンライン掲載されました。
背景
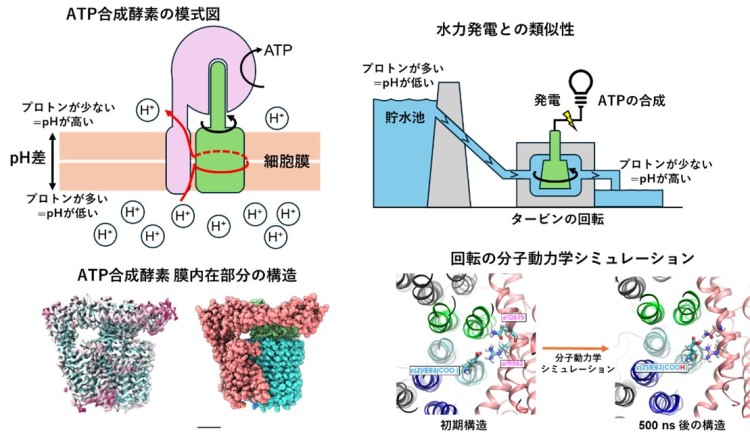
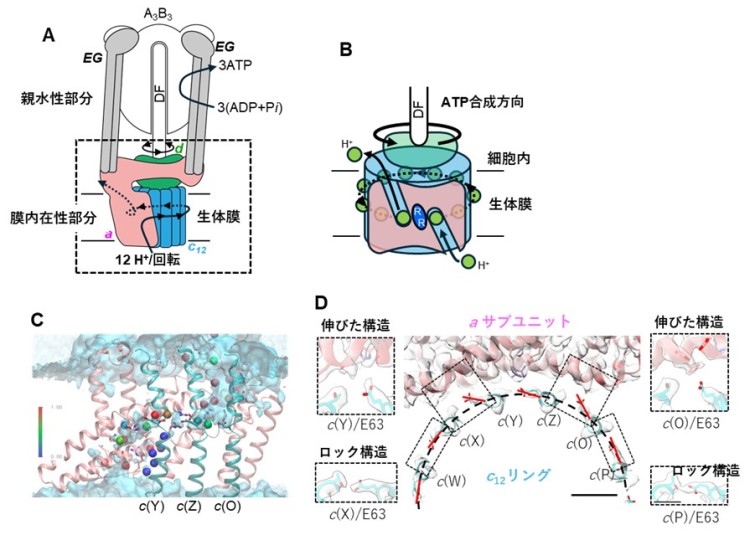
ATP合成酵素は、膜に埋まった膜内在性部分と膜から突き出た親水性部分からなります(図2)。膜内在性部分では、プロトン駆動力による水素イオンの移動により、リング構造が分子内部で回転し、親水性部分の軸部分がつられて回転することによりATPが合成されます(図2AB)。親水性部分でのATP合成反応の詳細については、これまでの研究によりほぼ解明されている一方で、膜内在性部分での水素イオンの流れによる回転機構は、その詳細な構造が不明なこともあり、未解決の課題として残されていました。本研究では、膜内在性部分における水素イオンの流れによる回転機構の解明を目的として、ATP合成酵素の膜内在性部分の詳細な構造を決定し、その構造情報に基づいた分子動力学シミュレーションを行いました。
研究手法・成果
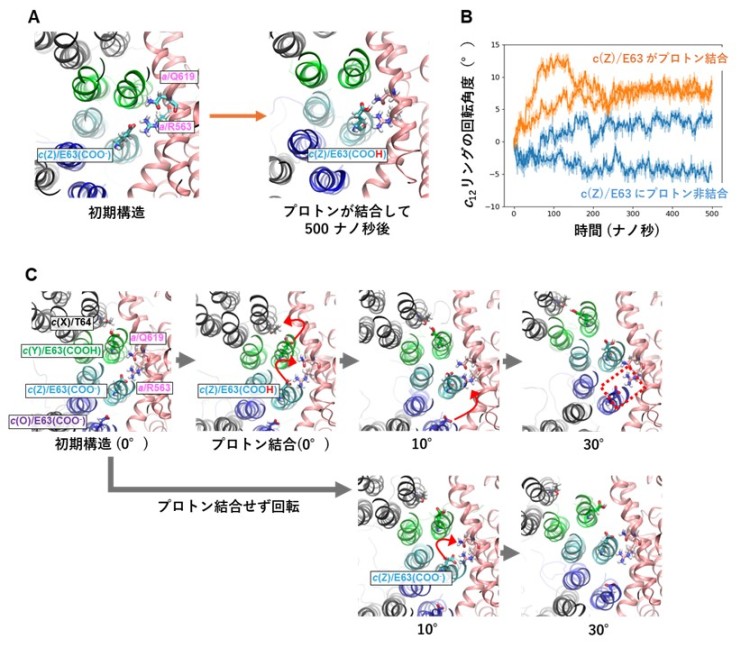
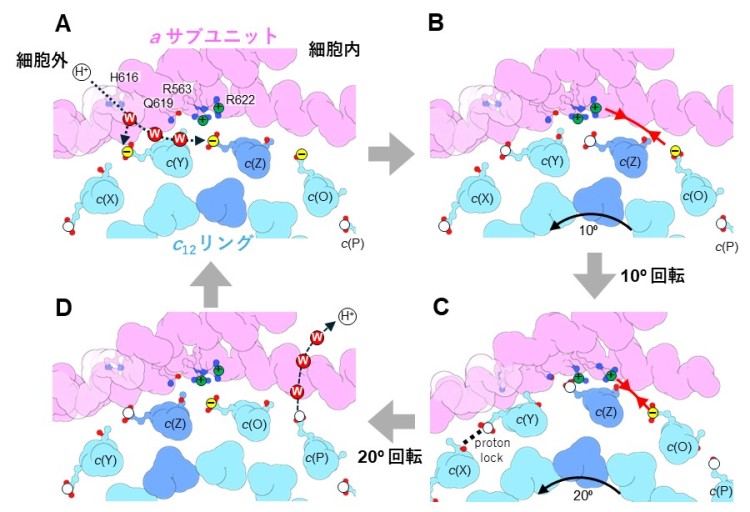
今回、私達は単離した膜内在性部分の構造を7)最大分解能2.8 Åで決定しました(図1)。その結果、膜内在性部分のaサブユニットとc12リングの間の親水的な隙間に複数の水分子を同定することができました(図2C)。水分子は、プロトンを輸送するうえで道筋となる重要な分子です。また、プロトンを輸送するために重要なc12リングのアミノ酸残基(63番目のグルタミン酸/E63)が、プロトンの結合状態によって向きがことなることを確認しました(図2D)。特に、親水的な隙間に存在している3つのE63は、水素イオンを離した電離状態になっており、真ん中の E63(c(Z))がaサブユニットのアルギニン残基と静電的に強く結合していることが確認されました。(図2D c(Y)、c(Z)、c(O))。そこで、その3つのE63の電離状態に着目し、分子動力学シミュレーションにより、3つのE63の電離状態が回転に及ぼす影響を検証しました。c(Z)のE63 を水素イオンが結合した状態にすると、シミュレーション中でc12リングがaサブユニットに対して、約10°回転しました(図3)。また、c(Z)のE63に水素イオンが結合しない状態で、c12リングを強制回転させると、リングと a サブユニット間の相互作用が強くなりスムーズにリングが回転しないことが示されました(図3)。また、c(Z)をはさむc(Y)、c(O)のE63のプロトン結合状態も回転に影響することもわかりました。これらの結果から、リングに存在する3つのE63 の荷電状態が水素イオンの流れによって不均一になり、そのためリングのブラウン運動の方向に偏りができ、リングが一方向に回転することが示唆されました(図4)。
波及効果・今後の予定
参考図
研究プロジェクトについて
用語・事項の解説
1. クライオ電子顕微鏡法 (cryo electron microscopy)
急速凍結したタンパク質などの生体試料に電子線を照射し、試料の観察を行うために透過電子顕微鏡を行う。得られた2次元像から画像解析により、3次元再構成を行い、生体分子の立体構造を決定する。
2. 分子動力学シミュレーション (Molecular dynamics simulation)
タンパク質の動きを計算する手法の1つ。タンパク質分子を原子レベルでコンピュータの中に再現し、原子間・分子間の相互作用に拘束を与えることで、タンパク質の動きを再現する。シミュレーションの細かさによって、QM/MM、全原子、粗視化などがある。
3. ATP (adenosine triphosphate)
様々な生体反応にエネルギー源として使われるため、細胞のエネルギー通貨とも呼ばれる。ヒトをはじめ多くの生物では、ATP合成のほとんどはATP合成酵素によって担われている。
4. ATP合成酵素 (ATP synthase)
ATPの合成を担うタンパク質。真核生物の場合、ミトコンドリア内膜、細菌の場合は細胞膜に存在する。膜に埋まった部分で、生体膜内外に形成されたpH差を回転に変換し、膜から突き出た部分で回転力を使ってATPを合成する(図1)。回転によって反応を行うため、回転分子モーターとも呼ばれる。
5. 好熱菌 Thermus thermophilus
静岡県伊豆の温泉で発見された至適生育温度が約75℃の好気性細菌。本菌由来のタンパク質は、熱耐性が高く、安定なため、構造解析に向いている。
6. 膜タンパク質 (Membrane proteins)
その一部、または全体が生体膜の中に埋まっているタンパク質。細胞内外の情報伝達や物質の輸送などに関わる。タンパク質全体の約3割が膜タンパク質である。7. 分解能(Resolution)、Å
分解能は、2つの点が2つの点として区別できる解像度を言う。数字が小さいほど、より高解像度の立体構造であることを示す。Åは、100億分の1メートル。構造解析分野において、解像度を示す単位として用いられる。
論文情報
| 論文タイトル | 「Rotary mechanism of the prokaryotic Vo motor driven by proton motive force.」 (原核生物Voモーターのプロトン駆動力による回転メカニズム) |
|---|---|
| 掲載誌 | Nature Communications |
| 掲載日 | 2024年11月20日(水)19:00(日本時間) |
| 著 者 | 【筆頭】Jun-ichi Kishikawa1,2,3, Yui Nishida1, Atsuki Nakano1, Takayuki Kato2, Kaoru Mitsuoka4, 【責任】Kei-ichi Okazaki5, 6* and Ken Yokoyama1* |
| 所 属 | 1Department of Molecular Biosciences, Kyoto Sangyo University 2Institute for Protein Research, Osaka University 3present affiliation; Department of Applied Biology, Kyoto Institute of Technology 4Research Center for Ultra-High Voltage Electron Microscopy, Osaka University 5Research Center for Computational Science, Institute for Molecular Science National Institutes of Natural Sciences 6Graduate Institute for Advanced Studies, SOKENDAI |
| DOI | 10.1038/s41467-024-53504-x |
- お問い合わせ先
-
京都産業大学 広報部
〒603‐8555 京都市北区上賀茂本山
Tel.075-705-1411
kouhou-bu@star.kyoto-su.ac.jp
