【生命科学部】クライオ電子顕微鏡によりATP合成酵素 FoF1 のスタートアップ機構を解明
2022.07.15
—謎に包まれていたATP合成酵素FoF1の初期反応機構(スタートアップ機構)をクライオ電子顕微鏡で解明。ATP合成酵素の分子機構の全容解明に一歩近づく—
生命科学研究科の中野 敦樹さん(博士課程1年次生)、横山 謙教授、および岸川 淳一さん(現大阪大学蛋白質研究所 助教)らのグループは、長年謎に包まれていたATP合成酵素の初期反応(ユニサイト触媒)で起こる現象を、クライオ電子顕微鏡で捉えることに成功しました。回転機構によるATP合成の全容解明への大きな一歩になります。この研究成果は、7月13日付けの PNAS Nexus電子版(Oxford 出版社)に掲載されました。
概要
私たちの生命活動は、高エネルギー物質であるATP(アデノシン三リン酸)によって支えられており、その大部分は、ATP合成酵素FoF1 により合成されています。FoF1は、またATPを分解することで回転する回転分子モータータンパク質でもあります。
FoF1 には ATP が結合する3つの触媒部位があり(図1)、すべての結合部位に ATP が結合した時に回転が連続的に起こります(定常状態)。しかし、最初のATPが1つ目の触媒部位に結合したときに起こる触媒反応(ユニサイト触媒)では、ATPに対する親和性が高く、かつその分解速度が遅いなど、特異な性質を示すことがわかっていました。多くの研究者が ユニサイト触媒機構の解明に取り組んできましたが、未だユニサイト触媒と定常状態での触媒機構との関連はわかっていませんでした。
京都産業大学の研究グループ(代表:京都産業大学 生命科学部 横山 謙教授)は、クライオ電子顕微鏡を用いた構造解析により、ユニサイト触媒の起こる様子を捉えることに成功しました。その結果、ユニサイト触媒は、定常状態とは異なる機構で起こる反応であり、ATPに対する高親和性は、ユニサイト触媒に特有な現象であることが示唆されました。今回の成果により、FoF1の分子機構の全容解明に一歩近づくことができました。
FoF1 には ATP が結合する3つの触媒部位があり(図1)、すべての結合部位に ATP が結合した時に回転が連続的に起こります(定常状態)。しかし、最初のATPが1つ目の触媒部位に結合したときに起こる触媒反応(ユニサイト触媒)では、ATPに対する親和性が高く、かつその分解速度が遅いなど、特異な性質を示すことがわかっていました。多くの研究者が ユニサイト触媒機構の解明に取り組んできましたが、未だユニサイト触媒と定常状態での触媒機構との関連はわかっていませんでした。
京都産業大学の研究グループ(代表:京都産業大学 生命科学部 横山 謙教授)は、クライオ電子顕微鏡を用いた構造解析により、ユニサイト触媒の起こる様子を捉えることに成功しました。その結果、ユニサイト触媒は、定常状態とは異なる機構で起こる反応であり、ATPに対する高親和性は、ユニサイト触媒に特有な現象であることが示唆されました。今回の成果により、FoF1の分子機構の全容解明に一歩近づくことができました。
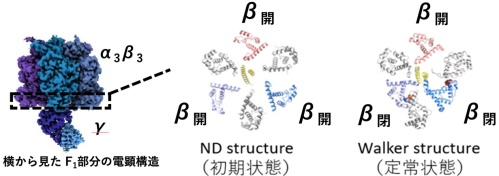
左: 横から見た F1部分のクライオ電子顕微鏡マップ。右:点線で囲った部分を下から見た図。触媒部位が存在する3つのβ(赤、紫、青)が、αを挟んで交互に並び、中心にγがある。ヌクレオチドを持たない初期状態(ND structure)では、3つのβが開構造をとる。Walker構造では、3つのβは、閉、閉、開構造をとる。
背景
私たち生命は、ATP(アデノシン三リン酸)を加水分解することによって得られるエネルギーで生命活動を維持しています。ほとんどの ATP は回転分子モーターである FoF1-ATPaseによって合成されます。FoF1 は、親水性のF1部分と膜内在性の F0 から構成されます。単離した F1 は、3つのαβからなるシリンダー構造 (α3β3)の中心に回転軸であるγサブユニットが刺さった構造からなります(図1参照)。F1部分単独でATPase 活性を持つことから F1-ATPase と呼ばれ、1970年頃から現在に至るまでF1-ATPaseを使った多くの研究が行われてきました。1994年にX線結晶構造解析により F1-ATPase の構造が明らかになりました。その構造では、3つの βサブユニット は、それぞれ異なったヌクレオチド結合状態をとり、1つには ATP が結合 (βTP)、もう一つには ADP(βDP)が結合、最後のものにはなにも結合していませんでした (βE)。そして、βTPとβDPは、閉じた構造、βE は、開いた構造になっていました。我々はこの構造を ”Walker 構造” と呼んでいます。定常状態での回転では、3つの β サブユニット上において、共同的にヌクレオチドの結合状態や構造を変化させることで、連続的にγサブユニットを回転させます。しかし、ATPが一つの触媒部位にしか結合できない条件 (ユニサイト条件)では、ATPに対して非常に高い親和性 (Kd = 10-12) を示し、しかも結合した ATPをゆっくりと加水分解することが報告されていました(ユニサイト触媒)。このユニサイト触媒機構と定常状態での触媒機構との関係を解明することは、回転分子モーターの分子機構を理解する上で必要です。しかし、1994年以降、定常状態での回転機構に対する研究が進む一方、ユニサイト 触媒の分子基盤は、わからないままでした。
結果
中野 敦樹(博士1年)、横山教授、岸川助教(現大阪大学)、中西 温子研究員らのグループは、クライオ電子顕微鏡を用いることにより、ユニサイト触媒が起こる前後の構造を捉えることに成功しました。クライオ電子顕微鏡による構造解析は、X線結晶構造解析で得ることのできなかった一過性の中間体構造を決定することができます。 透析処理によって内在性のヌクレオチドを取り除いた FoF1 を用い、FoF1の量に対して約1/3量のATP を加え、反応中に急速凍結しました。 得られた凍結グリッドをクライオ電子顕微鏡で撮影し、その画像を解析することで、すべての触媒サブユニットにヌクレオチドが結合していないND 構造と1つの触媒サブユニットにADP が結合したUS 構造 (ユニサイト構造)が得られました。ND構造は、Walker 構造 と大きく異なっており(図1)、3つのβ がすべて開いた構造をとっていました。 US構造では、βTP に ATP ではなく、加水分解後の ADP が結合していました。この結果から、ユニサイト触媒では、ATPはβDP ではなく、βTP に結合し、βTP で加水分解が起こるということが明らかになりました。以上のことから、FoF1 にヌクレオチドが結合していない初期状態から定常状態までのモデル(図2)を提案し、長年の議論に終止符を打つことができました。
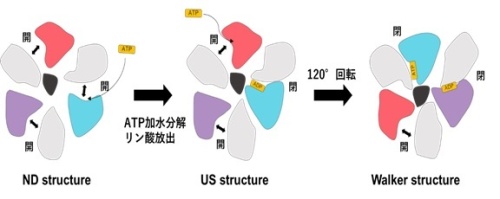
初期状態(ND structure)にATPが結合すると、1つのβが閉じ、加水分解、リン酸の放出まで起こったのがユニサイト構造(US structure)である。さらに、もう一つのATPが結合し、120°回転すると定常状態であるWalker構造に移行し、連続的に回転しながらATPを加水分解する。
今後の展開
本研究により、ユニサイト触媒の分子基盤、定常状態へと移行する過程を解明することができました。ATP駆動性の回転分子モーターの仕組みを理解するのに必要な重要なピースが埋まったといえます。このナノサイズで機能する分子機械の仕組みを理解することで、将来のナノマシーンの駆動部に応用できる人工分子モータータンパク質設計への応用が期待されます。
論文
| タイトル | 「Structural basis of unisite catalysis of bacterial FoF1-ATPase」 (FoF1 ATPase のユニサイト触媒機構の分子基盤) |
|---|---|
| 掲載誌 | PNAS Nexus電子版(Oxford 出版社) |
| 掲載日 | 2022年7月11日 |
| 著者 | 中野敦樹1 岸川淳一、中西温子、光岡薫、横山謙2 (1筆頭著者、2責任著者、) |
| DOI | https://doi.org/10.1093/pnasnexus/pgac116 |
謝辞
本研究は、科学研究費補助金(基盤研究(B))20H03231、20K06514、武田特定研究(武田財団)、京都産業大学 タンパク質動態研究所、感染分子研究センター、AMEDの創薬・生命科学研究支援プラットフォーム事業(BINDS)(助成番号1312)、文部科学省「ナノテクノロジープラットフォーム」の支援により行われました。
