生命科学研究科の西田 菜七実さん、羽深 典之研究員と津下 英明教授等のグループの研究が国際学術雑誌 Toxins (Basel)の学術論文として掲載されました。
京都産業大学 生命科学研究科の西田 菜七実さん、羽深 典之研究員と津下 英明教授等のグループの研究が国際学術雑誌 Toxins (Basel)の学術論文として掲載されました。これは、日本女子大学、大阪大学、大阪医科薬科大学と進めている共同研究の一部です。
オシロイバナ由来の抗ウイルスタンパク質MAPは、広く植物に存在するリボソーム不活性化タンパク質(RIP)の仲間で、大腸菌リボソームを構成するRNAから、特異的に1つのアデニン塩基*だけを引き抜いてく、リボソームの機能を損なわせる、N-グリコシラーゼ活性を持っています。本研究では、複数のMAP変異体を作製、生産精製することで、1)その分子構造を解明し、活性のメカニズムを推定しました。2)他のRIPとの比較から、リボソームの特異性のメカニズムを明らかにしました。
*大腸菌23SリボソームRNAの2660番目のアデニン(A2660)、その領域のRNAをサルシン・リシン ループ(SRL)と呼びます。
内容
野生型MAP(wt-MAP)は、その毒性のために大腸菌での発現ができません。このためN-グリコシラーゼの活性部位に存在すると考えられた2つのアミノ酸を置換して不活性化したMAP(E168Qと R171Qの2つの変異をもつMAP-EQRQ)を大腸菌で発現し、結晶化、その結晶構造を2.1Å分解能で明らかにしました。次にMAPの活性をRT-qPCRをもちいて測定しました。wt-MAPとMAP E168Q、MAP R171Q、MAP-EQRQで結果を見ると、その活性はMAP-EQRQとMAP R171Qで大きく減少しました。また、AlphaFold3を用いて、wt-MAP、MAP E168Q、MAP R171Q、MAP-EQRQのそれぞれと12ntRNAの複合体構造を予測して、その構造を比較しました。AlphaFold3では5つの構造が出力されますが、wt-MAPとSRLのRNAの複合体構造では、そのRNAのA2660がフリップした均一の構造に収束することがわかりました(図1)。興味深いことに、MAP R171Qでは 2塩基横のアデニンA2662が異なる構造でフリップしていること、MAP―EQRQではRNAの構造に多型が見られることが明らかになり、これらの知見からMAPのR171がSRLへのN-グリコシラーゼ活性で特に重要な役割をすることを明らかにしました。
さらに、リボソームとRIPの相互作用を明らかにしたいと考えています(図2)。また抗ウイルス活性との関連についても、今後の研究で明らかにしたいと考えています。
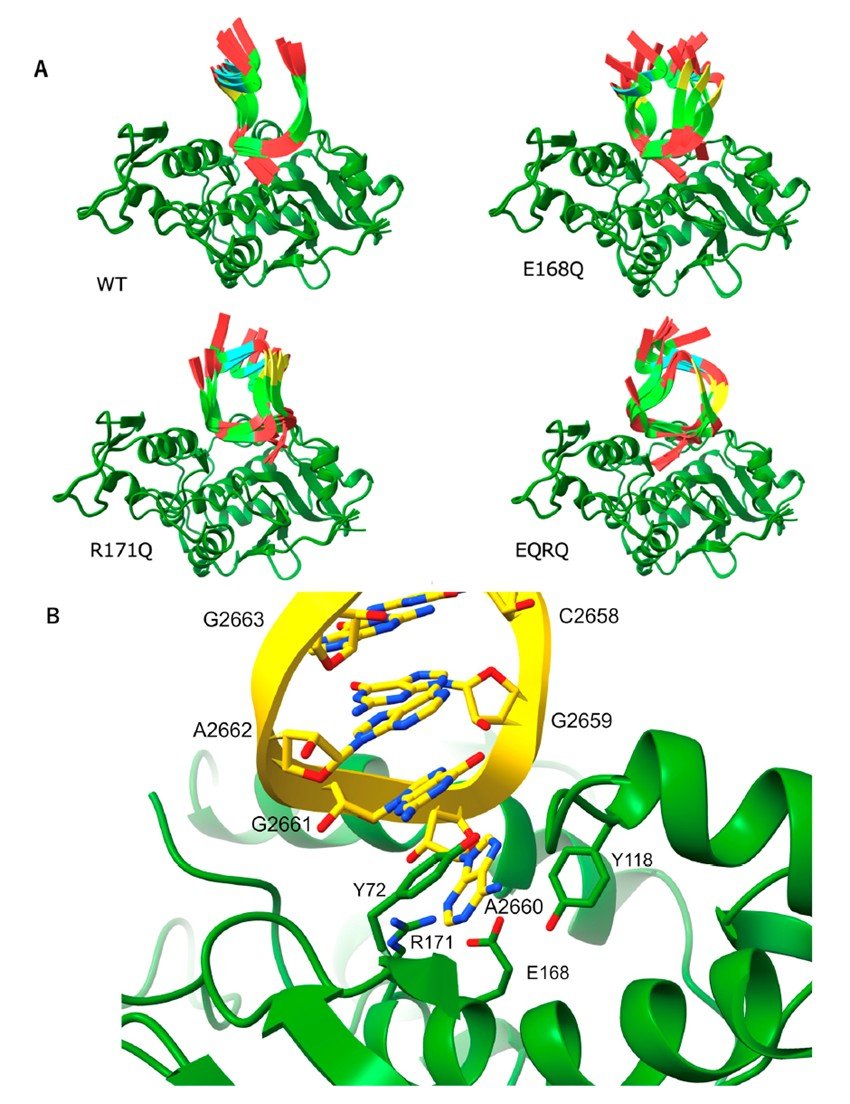
図1
A wt-MAP, MAP E168Q, MAP R171Q, MAP-EQRQとSRL12nt RNAとの複合体の予測構造(AlphaFold3) それぞれ5つの予測構造を重ねたもの。
RNA部分の構造に違いが見られる。
B wt-MAPとSRL 12nt RNAとの複合体の予測構造:どれもA2660がフリップアウトした均一な構造に収束した。このうちの1つの構造を示す。
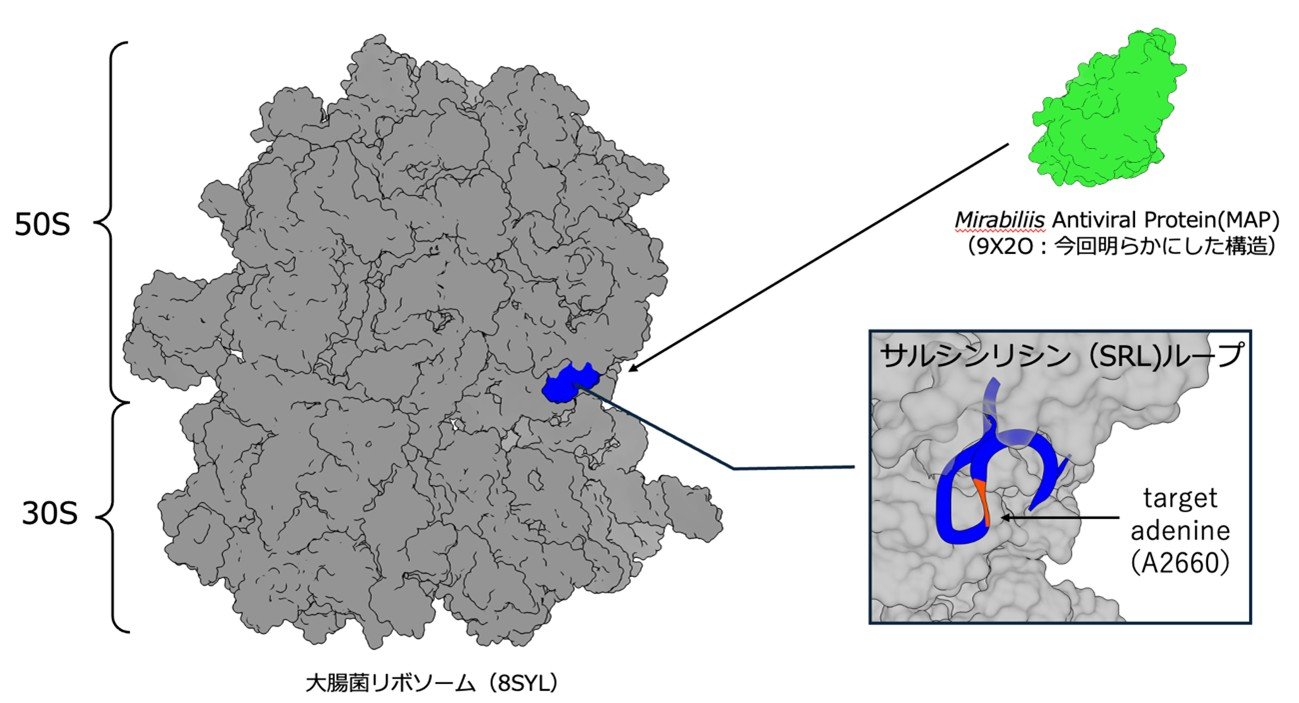
図2 MAPはいかにリボソーム サルシンリシン(SRL)ループを攻撃するか
論文情報
| タイトル | Structure of Ribosome-Inactivating Protein from Mirabilis jalapa and Its L12-Stalk-Dependent Inhibition of Escherichia coli Ribosome |
|---|---|
| 掲載誌 | Toxins |
| 掲載日 | 2025年11月28日 |
| 著者 | Nanami Nishida, Yuki Ninomiya, Hideaki Tsuge*, Noriyuki Habuka*(京都産業大学 生命科学部) Toru Yoshida (日本女子大学 理学部) Takehito Tanzawa (大阪大学 蛋白質研究所) Yasushi Maki, Hideji Yoshida (大阪医科薬科大学 医学部) *責任著者 |
| DOI | https://doi.org/10.3390/toxins17120575 |




